Frecuencia del polimorfismo C1858T del gen PTPN22 y marcadores de autoinmunidad en pacientes chilenos con diabetes tipo 1 y enfermedad celíaca
Francisca Salas-Pérez1, Tamara Loeff W.1,2, Carolina Pizarro A.1, Karla Vasquez O.1,
Elena Carrasco P.1,
Magdalena Araya Q.2 y Francisco Pérez-Bravo1,2
C1858T polymorphism of protein tyrosine phosphatase, non-receptor type 22 (PTPN22) gene in diabetic and celiac patients
1Laboratorio de Genómica
Nutricional. Departamento de
Nutrición. Facultad de Medicina.
Universidad de Chile.
2Laboratorio de Inmuno-genética.
Instituto de Nutrición y Tecnología
de los Alimentos (INTA). Universidad
de Chile.
Correspondencia a:
Dr. Francisco Pérez-Bravo
Laboratorio de Genómica Nutricional
Departamento de Nutrición
Facultad de Medicina
Universidad de Chile
Independencia 1027 (3º piso)
Santiago, Chile.
Teléfono: 56-2-978 6242
E-mail: fperez@med.uchile.cl
Recibido: 17 de enero de 2012
Aceptado: 15 de marzo de 2012
Background: A genetic polymorphism called C1858T of protein tyrosine phosphatase, non-receptor type 22 (PTPN22) gene has been associated with autoimmune diseases Aim: To describe the association between two autoimmune diseases, namely type 1 diabetes (T1D) and celiac disease (CD) and tyrosine phosphatase gene polymorphisms (variant C1858T of PTPN22). Subjects and Methods: C1858T single-nucleotide polymorphism within the PTPN22 gene was genotyped in 209 patients with T1D, 43 celiac patients and 100 healthy controls. Results: CC gene frequency was 0.906 and 0.790 in CD patients and controls respectively( p < 0.01). All analyzed groups had a low frequency of the TT genotype. Compared with the other study groups, patients with T1D had a low frequency of CC genotype (0.636). Also, in these patients, there was a non-significant association between CC genotype and islet cell IA-2 auto antibodies (p < 0.065). Among CD patients, CC genotype was significantly associated with anti-transglutaminase or anti endomysial antibodies (p < 0.03). Conclusions: These results confirm the association of the genetic variant C1858T of PTPN22 with CD. In contrast to published data, this association was not found in T1D patients.
Key words: Type 1 diabetes, genetic markers, Polymorphisms, PTPN22, Autoimmunity.
Introducción
La diabetes tipo 1 (DM1) es un trastorno autoinmune dirigido a la β-células en el páncreas resultante de un proceso autoinmune mediado por células T1. Corresponde a una de las enfermedades pediátricas más complejas desde la perspectiva epidemiológica y genética2,3. Muchos estudios y recientes revisiones han demostrado que la mayoría de los países muestra un incremento progresivo y sostenido de la DM1 en los últimos años4. La explosión epidemiológica de nuevos casos ha centrado la atención en los múltiples factores de riesgo involucrados tales como: la predisposición genética, la dieta, la estacionalidad, los agentes virales, la geografía, la autoinmunidad y la higiene ambiental5-8. Desde el punto de vista inmunológico, la presencia de autoanticuerpos contra epítopes pancreáticos como anticuerpos anti-insulina (AIA), la isoforma 65 kDa de la decarboxilasa del ácido glutámico, auto-anticuerpos (GAD65) y los anticuerpos asociados a insulinoma (IA-2) son indicadores altamente predictivos del riesgo para desarrollar DM1 cuando se presentan con títulos altos9. Se postula que la seroconversión podría ocurrir a edades tempranas (< 10 años de edad), sin embargo, se ha demostrado que la autoinmunidad puede ocurrir a cualquier edad10,11.
En relación a la enfermedad celíaca (EC), esta corresponde a una alteración inmune intestinal crónica con un fuerte componente genético. Su manifestación está gatillada por la ingestión de gluten, proteína que se encuentra en trigo, cebada y centeno. Las manifestaciones clínicas que la caracterizan son síntomas gastrointestinales, típicamente síndrome de mala absorción intestinal (presentación clínica clásica) y manifestaciones extraintestinales (presentaciones atípicas). Estas últimas a menudo son secundarias a un síndrome de mala absorción subclínico, como por ejemplo, episodios repetidos de anemia ferropriva u osteopenia y osteoporosis en edades tempranas, baja talla u otras en las que predominan los procesos autoinmunes12-14.
Desde el punto de vista genético, la DM1 y la EC comparten la gran mayoría de los marcadores genéticos clásicos, principalmente los marcadores HLA de clase II15. En Chile, la susceptibilidad genética de enfermos celíacos se ha asociado con la conformación DQ8. Esto es concordante con la alta frecuencia de los alelos DR4 (en desequilibrio del ligamiento con DQB1*0302) detectado en los grupos de Amerindios en Chile16,17. En otros países de América Latina como Argentina, se ha visto que un 95% de los pacientes celíacos son portadores de la conformación DQ2 versus un 40% en controles.
Tanto DQ2 como DQ8 corresponden a conformaciones HLA de clase II también muy frecuentes en población chilena con DM1 y con EC18.
Dentro de los marcadores genéticos no-HLA que se han estudiado tanto en la EC como en la DM1 se encuentra el gen de la tirosina fosfatasa linfoide (PTPN22). Este gen se ubica en el cromosoma 1p13 y codifica para la proteína tirosina fosfatasa linfoide también conocida como “Lyp”. La proteína Lyp inhibe la transducción de señales del receptor de células T (TCR) por desfosforilación de quinasas importantes para la señalización del receptor19. Existe un polimorfi smo genético denominado C1858T (Arg620Trp) en el gen PTPN22, el cual ha sido extensamente estudiado en diversas patologías autoinmunes20,21. La interacción que se produce en la región de Lyp que contiene el polimorfismo genético C1858T interactúa en forma distinta de acuerdo a la carga alélica del portador, lo que produce como resultado una mayor o menor eficiencia en la supresión de la señalización del TCR. Se postula que esta ganancia de función puede predisponer a la DM1 a través de un aumento de la supervivencia de las células T auto-reactivas durante la selección en el timo. Dado que la DM1 y la EC comparten algunos rasgos genéticos, pero no existe un conocimiento acabado de ellos, el propósito de este estudio fue estimar la frecuencia del polimorfi smo C1858T y evaluar la posible asociación entre este polimorfismo del gen PTPN22 y el componente autoinmune en estas dos patologías.
Pacientes y Metodología
El estudio fue realizado en Santiago, Chile con pacientes diabéticos tipo 1 diagnosticados a través de métodos estandarizados (WHO DiaMond Project) durante el período comprendido entre enero de 2007 a diciembre de 2010. Se incluyeron 209 casos diagnosticados durante este período (edad promedio 9,5 ± 4,2 años, rango de edad 4 a 17 años). La edad de diagnóstico de los pacientes con DM1 fue de 6,2 ± 3,5 años. Además se incluyeron 43 pacientes con EC diagnosticada a través de los criterios internacionales vigentes (que incluyen biopsia intestinal), con edad promedio 25,8 ± 8,5 años (rango de edad 15 a 41 años) y 100 controles, sin diabetes, ni antecedentes de patologías autoinmunes (edad promedio 14,8 ± 3,2 años, rango de edad 10 a 19 años). Las muestras de sangre pertenecientes a los pacientes diabéticos fueron obtenidas dentro de un período de tiempo cercano al debut (2-20 días). Todos los pacientes, controles y sus padres firmaron un consentimiento informado para participar en esta investigación. Este estudio fue aprobado por el Comité de Ética de la Facultad de Medicina, IDIMI e INTA de la Universidad de Chile.
Análisis de anticuerpos
Se realizó un screening serológico para los auto-anticuerpos en las muestras obtenidas de los pacientes DM1 y celíacos y controles). Los autoanticuerpos medidos fueron: anti-ácido glutámico descarboxilasa (65KD) (anti-GAD65) y anti-tirosina fosfatasa (anti-IA2), realizados mediante ELISA comercial (MEDIPAN, Medizin, Alemania). La determinación de anticuerpos anti-transglutaminasa (TTG) y anti-endomisio (EMA) se realizó en suero de pacientes celíacos, mediante ELISA, a través de un kit comercial (IMMCO DIAGNOCTICS, USA). Los coeficientes de variación intraensayo e interensayo para estas determinaciones estuvieron todas dentro de los rangos específicados por el fabricante (intraensayo < 2%; interensayo < 4%). La determinación de todos los anticuerpos se realizó en muestras de suero recolectadas con posterioridad al diagnóstico una vez que el paciente ya se encontraba estabilizado desde el punto de vista glicémico y con dosificación de insulina.
Análisis de polimorfismo genético
De cada paciente y control se obtuvo una muestra de sangre periférica de 3 ml. El suero sobrenadante y los residuos sólidos fueron separados por centrifugación. Se utilizaron 500 ml de sangre para obtener ADN genómico usando un protocolo estándar.
El polimorfismo C1858T se determinó utilizando un método previamente descrito22. Un fragmento de 218 pb que contiene la sustitución de nucleótido simple (SNP) se amplificó utilizando como partidor F1: 5`-ACTGATAATGTTGCTTCAACGG y el partidor reverso R1: 5`-TCACCAGCTTCCTCAACCAC. La reacción de amplificación se realizó con 20 ng de ADN genómico, buffer MgCl2, desoxirribonucleótidos trifosfato (dNTP) (1,25 mmol/L), partidores (20 pmol/ml), dimetil sulfóxido (DMSO) y Taq polimerasa. Las muestras fueron inicialmente desnaturalizadas durante 2 min a 94º C seguido por 35 ciclos de 94º C durante 30 s, 30 s en 60º C, y 30 s en 72º C, y una extensión final de 2 minutos a 72ºC. Los productos amplificados fueron digeridos con 10 unidades de RsaI (New England Biolabs, MA, EE.UU.) en una reacción de 3 horas a 37º C. Los productos digeridos fueron sometidos a electroforesis en un gel de agarosa al 2,5% y visualizados a la luz UV con bromuro de etidio. El alelo 1858T mutante no puede ser digerido y produjo un fragmento único de 218 pb, mientras que para la presencia del alelo 1858C se obtuvieron dos fragmentos, de 176 pb y 46 pb.
Análisis estadístico
Las comparaciones de frecuencia alélicas y genotípicas
entre casos y controles fueron analizadas mediante las pruebas
de x2 y Prueba exacta de Fisher utilizando el programa SHESIS
(URL: http://202.120.7.14/analysis/myAnalysis.php). Se
evaluó la concordancia de las frecuencias genotípicas con respecto al equilibrio de Hardy-Weinberg mediante la prueba
exacta de x2. Las comparaciones de las variables continuas del
estudio se realizaron mediante prueba t de Student y la comparación
del perfil de anticuerpos se realizó a través de análisis
de proporciones. Un valor de p menor de 0,05 fue considerado
como estadísticamente significativo.
Resultados
La Tabla 1 muestra las características de los pacientes
con DM1 y EC desde el punto de vista de su edad de diagnóstico,
IMC y porcentaje de autoanticuerpos. No se observan
diferencias al comparar los grupos y el título positivo de anticuerpos,
como se observa en la tabla alcanza al 75,7% entre
los pacientes con DM1 y al 45,8% en los pacientes con EC.
La población control fue negativa para los cuatro anticuerpos
analizados.
La Tabla 2 resume la información relacionada a la distribución genotípica y alélica de la variante C1858T del gen PTPN22 en los tres grupos estudiados. En todos los grupos se observa una muy baja frecuencia del genotipo TT, fenómeno que es común en todas las series publicadas para este gen en diversas poblaciones y grupos étnicos. Se observó una elevada frecuencia del genotipo CC en pacientes con EC (90,6%), lo que se encuentra dentro de las frecuencias reportadas en la literatura. Respecto al grupo con DM1, se observó una menor frecuencia para este genotipo, lo cual difiere de los trabajos publicados para la variante C1858T en pacientes con DM1, principalmente de origen caucásico. La distribución de frecuencias genotípicas en el grupo control, se situó dentro de los valores descritos en individuos sin patologías autoinmunes.
Tabla 1. Características clínicas y antropométricas de ambos grupos
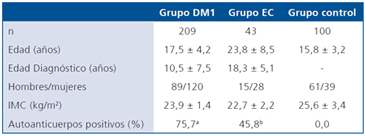 aGAD65 e IA-2. bTTG y EMA.
aGAD65 e IA-2. bTTG y EMA. Tabla 2. Distribución de alelos y genotipos para el polimorfi smo C1858T del gen PTPN22 en pacientes con DM1, pacientes celíacos y controles
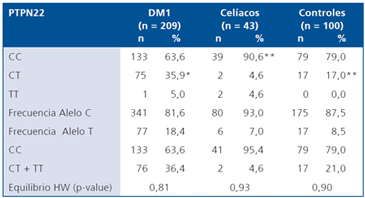
*p < 0,001 genotipo CT DM1 versus controles; **p < 0,036 celíacos versus controles (ambos genotipos).
La Tabla 3 presenta la información relacionada al perfil de autoinmunidad en pacientes con DM1 y celíacos en relación al perfil de auto-anticuerpos característicos en cada patología. Para anti-GAD65 en el grupo con DM1, no se observó una distribución diferente entre los portadores del genotipo CC versus los portadores del genotipo CT. Para la distribución del auto-anticuerpo IA-2, existe una tendencia de mayor frecuencia de pacientes positivos para este anticuerpo en el grupo portador del genotipo CC (68,4% versus 51,3%; p = 0,065). En el caso de la EC, se observó una mayor frecuencia de anticuerpos TTG y EMA en los individuos portadores del genotipo CC (p < 0,01).
Tabla 3. Asociación entre genotipos PTPN22 y perfi l autoinmune en pacientes con DM1 y EC
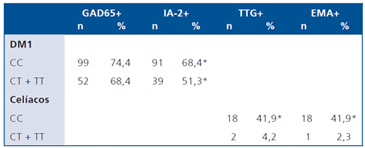
*p = 0,065 CC versus CT + TT en pacientes con DM1; p < 0,01 CC versus CT + TT en pacientes celíacos.
Discusión
Este estudio confirma la posible asociación del gen PTPN22/LYP con DM1. La variante alélica T expresada como la combinación CT + TT es significativamente más frecuente en los pacientes con DM1, en comparación con pacientes con EC y controles. Esta asociación no se observa para pacientes con EC, como ha sido publicado en otros estudios23.
A pesar de que esta asociación entre PTPN22 y DM1 ha sido consistente en diversos estudios poblacionales, lo observado en población chilena con EC no parece seguir el mismo curso. La comparación de frecuencias alélicas para el allelo 1858T es extremadamente baja comparada con el antecedente publicado. El papel de esta variante genética fue abordado por primera vez por Onengut-Gumuscu y cols24 quienes secuenciaron la región codificante del gen PTPN22 e identificaron varias variantes nuevas. El análisis de haplotipos sugirió que el alelo de riesgo 1858T está contenido en un solo haplotipo fuertemente asociado a DM1. En población asiática el sitio 1858 no es polimórfico, lo que podría dar cuenta de la baja frecuencia encontrada en nuestra población. En japoneses y coreanos, otro polimorfismo de nucleótido simple (SNP) 21123G/C ha demostrado estar débilmente asociado con DM1. En grupos caucásicos, este SNP también se asocia con DM119,23,25.
La DM1 y la EC son enfermedades multifactoriales, con una contribución genética importante. En ambas patologías, existe un amplio número de genes involucrados, siendo el complejo de histocompatibilidad mayor (HLA) de clase II el que presenta la más fuerte asociación en ambos casos, contribuyendo con un OR sobre 4, tanto para DM1 como para EC23. Dentro de los genes no HLA, ambas enfermedades comparten algunos genes de predisposición, tales como CTLA-4 y PTPN22, pero con contribuciones mucho menores (OR = 1,5).
Este estudio no mostró una asociación fuerte entre la presencia del polimorfismo PTPN22 y el perfil de autoinmunidad en pacientes con DM126,27. Se observó una mayor tendencia de asociación entre el genotipo CC con una mayor frecuencia de autoanticuerpos IA-2 positivos. Fenómeno similar al observado con otros anticuerpos en un reciente estudio de Kordonouri y cols, en el año 201026. De forma similar, el genotipo CC mostró mayor asociación con anticuerpos característicos de la EC. Sin embargo, la asociación descrita en otros estudios para PTPN22 y EC28, no se observó en nuestro análisis. En términos de frecuencia alélica para la variante menos frecuente (alelo T), las publicaciones europeas19 muestran un claro gradiente este-oeste con cifras que van desde un 15,5% en el norte de Europa (países nórdicos), frecuencias intermedias en Francia y España (7,8%) y muy baja frecuencia en Italia (2,5%). Paradojalmente, en DM1 nuestros datos de frecuencia alélica muestran valores de un 18% para el alelo T, frecuencia bastante alta si lo comparamos con las frecuencias observadas en pacientes celíacos y controles (7 y 8,5% respectivamente).
El polimorfismo C1858T del gen PTPN22 ha sido considerado el tercer locus de asociación para DM1, sin embargo, en nuestra población de pacientes con EC y DM1 parece cumplir un papel de menor importancia. Los estudios de polimorfismo genético involucrados en la etiopatogenia de la DM1 y al EC han incrementado significativamente nuestro conocimiento de las vías del sistema inmune que se ven afectadas en estas patologías. Es probable que una combinación de genes relacionados con el sistema HLA, sumado a los efectos causados por otros genes de menor impacto como la variaciones genéticas en PTPN22 y CTLA-4, permitan aproximarse al riesgo potencial de desarrollar ambas enfermedades.
Agradecimientos
Este estudio ha sido parcialmente financiado por el PROYECTO FONDECYT Nº 1100075.
Referencias
- Morran MP, Omenn GS, Pietropaolo M. 2008. Immunology and genetics of type 1 diabetes. Mount Sinai J Med 75: 314-327.
- Concannon P, Rich S, Nepom GT. 2009. Genetics of type 1A
diabetes. N Engl J Med 360: 1646-1651.
- Van Belle TL, Coppieters KT, von Herrath MG. 2011. Type 1
diabetes: etiology, immunology and therapeutic strategies. Physiol
Rev 91: 79-118.
- Vehik K, Dabelea D. 2011. The changing epidemiology of type 1
diabetes: why it is going through the roof?. Diab Metab Res Rev
27: 3-13.
- Norris JM, Barriga K, Klingensmith G, Hoffman M, Eisenbarth
GS, Erlich HA, Rewers M. 2003. Timing of initial cereal exposure
in infancy and risk of islet autoimmunity. JAMA 290: 1713-1720.
- Lefebvre D, Powell K, Strom A, Scott F. 2006. Dietary Proteins as
Environmental Modifiers of Type 1 Diabetes Mellitus. Ann Rev
Nutr 26: 175-202.
- van der Werf N, Kroese F, Rozing J, Hillebrands JL. 2007. Viral
infections as potential triggers of type 1 diabetes. Diab Metab Res
Rev 23: 169-183.
- Gale EA. 2002. A missing link in the hygiene hypothesis?
Diabetologia 45: 588-594.
- Colman PG, McNair PD, Gellert S, Kewming K, Schmidli RS,
Steele CE, Harrison LC. 2002. Development of autoantibodies to
islet antigens during childhood: implications for preclinical type 1
diabetes screening. Pediatr Diabetes 3: 144-148.
- Orban T, Sosenko JM, Cuthbertson D, Krischer JP, Skyler JS,
Jackson R, et al. 2009. Pancreatic islet autoantibodies as predictors
of type 1 diabetes in the Diabetes Prevention Trial-Type 1. Diab
Care 32: 2269-2274.
- Litherland SA.2008. Immunopathogenic interaction of
environmental factos triggers and genetic susceptibility in
diabetes: is epigenetic the missing link?. Diabetes 57: 3184-3185.
- Fasano A. 2005. Clinical presentation of coeliac disease in the
pediatrics population. Gastroenterology 128: S68-S73.
- Green P. 2005. The many faces of coeliac disease: Clinical
presentation of celiac disease in the adult population.
Gastroenterology 128: S74-S78.
- Fasano A. 2011. Zonulin and its regulation of intestinal barrier
function: the biological door to immflamation, autoimmunity and
cancer. Physiol Rev 91: 151-175.
- Trynka G, Wijimenga C, van Hell DA. 2010. A genetic perspective
in celiac disease. Trends Mol Med 16: 537-550.
- Pérez-Bravo F, Araya M, Mondragón A, Ríos G, Alarcón T,
Roessler JL, Santos JL. 1999. Genetic differences in HLA-DQA1*
and DQB1* allelic distributions between celiac and control
children in Santiago, Chile. Hum Immunol 60: 262-267.
- Pérez-Bravo F, Martínez-Laso J, Martín-Villa JM, Moscoso J,
Moreno A, Serrano-Vela JI, Zamora J, et al. 2006. HLA non-class
II genes may confer type I diabetes susceptibility in a mapuche
(Amerindian) affected family. European Journal of Medical
Genetics 49: 37-41.
- Araya M, Mondragón A, Pérez-Bravo F, Roessler JL, Alarcón T,
Ríos G, Bergenfreid C. 2000. Celiac disease in a chilean
population carrying amerindian traits. J Pediatr Gastroenterol Nutr
31: 381-386.
- Burn GL, Svensson L, Sánchez-Blanco C, Saini M, Cope AP.
2011. Why is PTPN22 a good candidate susceptibility gen for
autoimmune diseases?. FEBS Lett 585: 3689-3698.
- Bottini N, Vanq T, Cucca F, Mustelin T. 2006. Role of PTPN22 in
type 1 diabetes and other autoimmune diseases. Semin Immunol
2006; 18: 207-213.
- Smyth DJ, Plagnol V, Walker NM, Cooper JD, Downes K,
Yang JH, et al. 2008. Shared and distinct genetic variants in type 1
diabetes and celiac disease. N Engl J Med 359: 2767-2777.
- Steck AK, Liu SY, McFann K, Barriga KJ, Babu SR, Eisenbarth
GS, et al. 2006. Association of the PTPN22/LYP gen with type 1
diabetes. Pediatr Diabetes 7: 274-278.
- Lee YH, Rho H, Choi SJ, Ji JD, Song GG, Nath SK, Harley JB.
2007. The PTPN22 C1858T functional polymorphism and
autoimmune diseases-a meta-analysis. Rheumatology 46: 49-56.
- Onengut-Gumuscu S, Ewens KG, Spielman RS, Concannon P.
2004. A functional polymorphism (1858C/T) in the PTPN22 gene
is linked and associated with type I diabetes in multiplex families,
Genes Immun 5: 678-680.
- Eyre S, Hinks A, Bowes J, Flynn E, Martin P, Wilson AG, et al.
2010. Overlapping genetic susceptibility variants between three
autoimmune disorders: rheumatoid arthritis, type 1 diabetes and
coeliac disease. Arthritis Research 12: R175.
- Kordonouri O, Hartmann R, Badenhoop K, Kahles H, Illonen J.
2010. PTPN22 1858T allele is associated with younger age at
onset of type 1 diabetes and is not related to subsequent thyroid
autoimmunity. Hum Immunol 71:731-732.
- Lempainen J, Vaarala O, Makela M,Veijola R, Simell O, Knip M,
et al. 2009. Interplay between PTPN22 C1858T polymorphism and
cows milk formula exposure in type 1 diabetes. J Autoimmunity
33: 155-164.
- Santin I, Castellanos-Rubio A, Aransay AM, Castaño L, Vitoria JC, Bilbao JR. 2008. The functional R620W variant of the PTPN22 gene is associated with celiac disease. Tissue Antigens 71: 247-249.